基于多聚腺嘌呤的二嵌段寡核苷酸探针用于卡那霉素 可视化检测
单链DNA(Single-stranded DNA,ssDNA)在未修饰的裸金纳米颗粒(AuNPs)上的吸附已成为基于核酸分子的比色分析的常用机制。本研究设计了一段包含多聚腺嘌呤(polyA 20)尾部序列的二嵌段寡核苷酸探针(dP),构建了一种免标记的AuNPs比色生物传感器用于检测卡那霉素(Kanamycin, Kana)。在卡那霉素存在的情况下,磁珠@双链DNA(MBs@dsDNA)复合物上的卡那霉素适配体序列(Kana-Apt)与其特异性结合,导致dP从磁珠表面释放。由于polyA 20与AuNPs之间有强吸附亲和力,dP被吸附在AuNPs表面,从而在高 盐环境中保护AuNPs不发生团聚。与具有相同碱基数量的随机序列(Random sequence,RS)相比,利用含有 polyA 20尾部序列的AuNPs比色策略对卡那霉素的检测灵敏度提高了2个数量级。此比色传感器对卡那霉素的线性检测范围为 0.01~1 nmol/L,检出限(LOD, S/ N=3)为 0.01 nmol/L。本方法无需大型仪器及其它信号放大策略,具有较高的灵敏度和特异性,适用于现场大批量样品的快速、可视化和实时检测,为其它抗生素以及影响食品安全的化学污染物的可视化检测提供了新思路。
卡那霉素(Kanamycin,Kana)是一种用于预防和治疗微生物感染的广谱氨基糖苷类抗生素,被用作农业和畜牧业中的饲料添加剂和兽药 [1] 。然而,滥用卡那霉素的现象屡见不鲜,导致其在动物源性食品中残留, 进而经由食物链进入人体,通过积累效应危害公众健康 [2] 。我国国家标准《食品安全国家标准 食品中农药最大残留限量》(GB 31650—2019) [3] 规定了不同食品中卡那霉素的最大残留限量(Maximum residue limits,MRLs),如牛奶中的MRL为 150 μg/kg,肌肉的MRL为 100 μg/kg。卡那霉素的检测方法主要包括色谱法 [4] 、酶联免疫吸附分析法 [5] 和毛细管电泳法 [6] 等。但是,大多数方法需要专业的操作人员和高精度仪器的支持,在现场实时检测和大批量筛查中的应用仍存在局限性。因此,建立简单、灵敏和低成本的卡那霉素检测方法对于保障食品安全和公众健康具有重要的现实意义。
目前,基于新型纳米材料构建的低成本、易小型化的生物传感器受到了广泛关注 [ 7- 8] 。其中,比色生物传感器因其具有操作简单、成本低和可视化识别等特性而备受关注,在大批量样品的筛查方面显示出良好的应用潜能。金纳米颗粒(Gold nanoparticles,AuNPs)的合成简单且易于生物功能化,常作为比色生物传感器的关键元件 [ 9- 10] 。此外,AuNPs表现出与粒子间距相关的表面等离子体特性 [ 11- 12] ,为比色法的现场应用提供了优良的纳米检测平台。值得注意的是,单链DNA(Single-stranded DNA,ssDNA)和双链DNA(Double-stranded DNA,dsDNA)在未经修饰的AuNPs上具有不同的吸附特性。由于ssDNA具有柔性,更易吸附在AuNPs上,而dsDNA的刚性双螺旋结构使其在AuNPs表面的吸附较差 [13] 。基于此原理的AuNPs比色传感策略已被广泛应用于许多领域,特别是食品安全现场筛查和个人医疗保健中的即时检测(Point-of-care testing,POCT) [ 14- 15] 。尽管该类免标记比色法简便快速,但其主要依赖于ssDNA对AuNPs的吸附亲和力,导致检测灵敏度受限。不同的DNA碱基与AuNPs表面相互作用的方式不同 [16] 。 已有研究证实,相比于随机序列的DNA,多聚腺嘌呤(Poly-adenine,polyA)对AuNPs表现出更高的吸附亲和力 [ 17- 18] , 这种独特性质使得polyA在生物分子编程、生物传感器和新型药物递送等研究领域得到了广泛应用 [ 19- 21] 。 然而,基于polyA和AuNPs之间高吸附亲和力的比色分析研究仍然较少 [22] 。
在传统AuNPs比色策略的基础上,本研究构建了一种基于二嵌段寡核苷酸探针(Diblock oligonucleotide probe,dP)的免标记比色适配体传感器用于卡那霉素的检测。与含相同碱基数量的随机序列(Random sequence,RS)相比,在不依赖任何信号放大技术的情况下,利用含有polyA 20尾部序列的AuNPs比色策略对卡那霉素的检测灵敏度提高了2个数量级,线性检测范围为 0.01~1 nmol/L,检出限为 0.01 nmol/L,并可实现裸眼检测,具有良好的特异性。本方法克服了比色传感器检测灵敏度低的问题,实现了卡那霉素的可视化灵敏检测。
1 实验部分
1.1 仪器与试剂
UV-2540紫外可见分光光度计(日本岛津公司);LQ-5103A型电子天平(上海瑶新电子科技有限公司);Mixer 4K微型涡旋混合仪(生工生物工程(上海)股份有限公司);HCM100-Pro 恒温振荡金属浴(大龙兴创实验仪器(北京)有限公司);D1008掌上离心机(大龙兴创实验仪器(北京)有限公司);琼脂糖凝胶成像系统(美国Bio-Rad公司);Bio-Mag MultiSep磁性分离器(美国Polysicences公司);9600基因扩增仪(珠海黑马医学仪器有限公司);78-1磁力加热搅拌器(江苏科析仪器有限公司)。
四氯金酸水合物(HAuCl 4·H 2O,99%,上海泰坦科技股份有限公司);二水柠檬酸三钠(Na 3C 6H 5O 7·2H 2O, 国药集团化学试剂有限公司);戊二醛溶液(Glutaraldehyde)、磁珠吡啶洗涤液(Pyridine Wash Buffer,PWB,1mol/L)、8600-10氨基磁珠(Magnetic beads,MBs, 50 mg/mL)购自美国Polysicences公司;20×磷酸盐缓冲液(Phosphate buffer saline,PBS,pH 7.4~7.6)、NaH 2PO 4、Na 2HPO 4、50×TAE缓冲液 (2 mol/L Tris-acetic acid, 100 mmol/L EDTA,pH 8.4)、琼脂糖III(TM)、4S Red Plus 核酸凝胶染色剂(10000×水溶液, 0.1 mL)、10×DNA上样缓冲液(DNA loading buffer)、NaCl、Kana、硫酸链霉素(Streptomycin sulfate, STR)、氧氟沙星(Ofloxacin,OFX)、恩诺沙星(Enrofloxacin,ENR)、盐酸林可霉素(Lincomycin hydrochloride, LIN)和诺氟沙星(Norfloxacin,NOR)购于生工生物工程(上海)股份有限公司。所用化学试剂均为分析纯;实验用水为Milli-Q 超纯水(美国Barnstead 公司超纯水仪制备)。实验所用的DNA 均购自生工生物工程(上海)股份有限公司,具体序列信息见 表1。DNA 干粉用超纯水配制成 100 μmol/L的储备液,然后用1×PBS (NaH 2PO 4-Na 2HPO 4,10 mmol/L)稀释至实验所需浓度。
表1 实验中所用的 DNA 序列
|
名称 Name |
碱基序列 Sequence (5′→3′) |
|
卡那霉素适配体 Kanamycin aptamer (Kana-Apt) |
NH 2-AAAAAATGGGGGTTGAGGCTAA GCCGAGTCAC |
|
互补序列-polyA 20 Hybridization block-polyA 20 (HB-polyA 20) |
AAAAAAAAAAAAAAAAAAAA GTGACTCGGC |
|
互补随机序列 Hybridization block-random sequence (HB-RS) |
CTAGCATGTGCATAGCTACGGTGACTCGGC |
|
互补序列-polyA 5 Hybridization block-polyA 5 (HB-polyA 5) |
AAAAAGTGACTCGGC |
|
互补序列-polyA 10 Hybridization block-polyA 10 (HB-polyA 10) |
AAAAAAAAAAGTGACTCGGC |
|
互补序列-polyA 30 Hybridization block-polyA 30 (HB-polyA 30) |
AAAAAAAAAAAAAAAAAAAAAAAAAAAAAAGTGACTCGGC |
注:下划线部分是互补的碱基序列。
Note: The underlined parts are complementary sequences.
1.2 实验方法
1.2.1 AuNPs 的制备
通过柠檬酸三钠还原氯金酸的方法合成AuNPs [23] 。将 50 mL超纯水和 50 μL 1%氯金酸溶液于磁力搅拌下混合,并加热至沸腾,迅速加入 1.75 mL 1%现配的柠檬酸三钠溶液,继续搅拌加热反应 30 min后,缓慢降至室温。产物于4 ℃避光保存。
1.2.2 MBs@Kana-Apt 的制备
按照产品说明书对 50 mg/mL 1.5 μmol/L的磁珠进行活化,将磁珠上的氨基转化为醛基。取 20 μL 醛基化磁珠,磁分离后加入 20 μL 10 μmol/L氨基修饰的Kana-Apt(预先于 90 ℃变性处理 5 min), 37 ℃振荡孵育过夜后进行磁分离,用 100 μL 1×PBS缓冲溶液充分洗涤磁珠,重悬于 20 μL 1×PBS中,得到的MBs@Kana-Apt体系于 4 ℃保存备用。
1.2.3 MBs@dsDNA 的制备
取 20 μL 10 μmol/L HB-polyA 20加入到 20 μL MBs@Kana-Apt中,混匀,于 90 ℃加热变性处理5 min, 37 ℃振荡孵育 2 h。磁分离后,用 100 μL 1×PBS洗涤5次,重悬于 20 μL 1×PBS中,得到的MBs@dsDNA体系于 4 ℃保存。
1.2.4 盐浓度的优化
在 30 μL超纯水中加入 60 μL AuNPs, 37 ℃恒温条件下避光孵育 2 h, 再加入不同浓度的NaCl溶液(0、0.1、0.2、0.3、0.4和 0.5 mol/L), 静置 1 min, 裸眼观察溶液颜色变化,并测定体系在520和 620 nm波长处的吸光度。
1.2.5 基于 polyA 的比色传感器用于卡那霉素检测
将 20 μL MBs@dsDNA和 30 μL不同浓度的卡那霉素(0、0.01、0.05、0.1、0.5、1、5、10、50、 100、500 nmol/L及 15 μmol/L)充分混匀后,室温孵育过夜。磁分离,移取 30 μL上清液,加入 60 μL AuNPs, 37 ℃避光孵育 2 h 后,加入 10 μL 0.3 mol/L NaCl溶液。静置 1 min后,裸眼观察颜色变化,并测定体系在520和 620 nm 波长处的吸光度。
1.2.6 特异性检测
20 μL MBs@dsDNA 与 30 μL不 同浓度的抗生素(卡那霉素、STR、OFX、LIN、ENR和NOR,卡那霉素的浓度为 10 nmol/L, 其它抗生素的浓度为 1 μmol/L)充分混匀后,室温孵育过夜。后续操作与1.2.5节 方法相同。
1.2.7 PolyA 长度的影响
设计不同长度的A碱基尾部序列探针(polyA 5、polyA 10、polyA 20和polyA 30)。将 20 μL MBs@dsDNA 和 30 μL不同浓度的卡那霉素(0、0.01、0.05、0.1、0.5、1、5、10、50、100和 500 nmol/L)充分混匀后,室温孵育过夜,后续操作与1.2.5节方法相同。
2 结果与讨论
2.1 实验原理
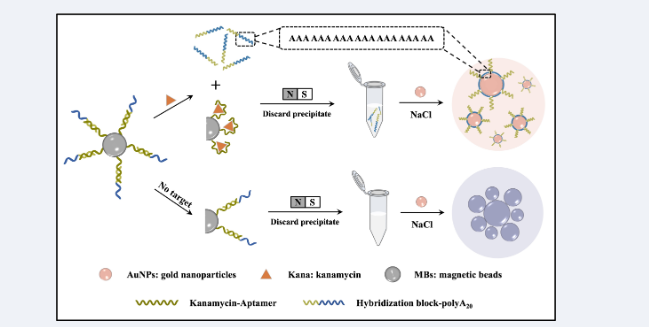
基于二嵌段寡核苷酸探针的比色适配体传感器用于检测卡那霉素的原理如 图1所示,此探针由polyA 20 和HB序列组成。首先,氨基化修饰的卡那霉素适配体Kana-Apt与醛基化磁珠发生反应后,HB序列与Kana-Apt互补配对,从而得到MBs@dsDNA复合物。MBs@dsDNA 复合物表面双链结构中的Kana-Apt与卡那霉素的亲和力更高,二者发生特异性结合,导致dsDNA解体,释放出dP。磁分离提取上清液中游离的dP与未经修饰的AuNPs溶液孵育,polyA 20与AuNPs表面发生高亲和力吸附。 AuNPs在polyA 20的保护下免受高盐浓度环境引发的聚集,溶液仍然保持红色。当体系中不含有卡那霉素时,MBs@dsDNA 结构不发生变化,在高盐条件下,AuNPs发生团聚,导致溶液体系由红色变为蓝灰色。
